Points of View ゲノム情報を研究利用する際の情報の流れ
医薬産業政策研究所 主任研究員 岡田法大
個人情報保護のための技術的措置の重要性
第211回国会において改正次世代医療基盤法が可決・成立し、新たに「仮名加工医療情報」の利用が始まる。従来の次世代医療基盤法では、医療情報を匿名加工することにより医薬品の研究開発等に利用することが可能となっていたが、今回新たに、仮名加工を行った医療情報も利用可能となる。仮名加工医療情報とは、他の情報と照合しない限り、個人を特定できないよう加工した情報と定義され、削除やカテゴリ化などの匿名加工が必要となっていた特異な臨床検査値なども研究に利用可能となる1)。また、審査当局による真正性確認のための情報照会が可能となることにより、薬事承認の申請等にも情報を利用できるようになる。仮名加工医療情報の研究利用は、法改正の議論の過程で製薬産業からも要望が出されていたものであり2)、製薬産業における医療情報利用を後押しするものとなることが期待される。
一方で、今回の改正が検討された次世代医療基盤法検討ワーキンググループでの議論や国会審議の過程では、仮名加工された医療情報を受領し、管理することとなる認定利用事業者に対して、情報の安全管理措置や、組織内部における利用部門の制限、個人情報の再識別に対する規制の強化等に関する意見が複数挙げられており、仮名加工医療情報を利用する場合、利用者の情報管理の負担は増加することが見込まれる。個人情報の保護の観点では、最も機微な生体情報であるゲノム情報の研究利用の体制に目を向けると、クラウドやコンテナ型の仮想化技術を利用し、情報の漏洩や不正利用に対するデータガバナンスの強化が進められている3)。これらの取り組みは、情報利用者に課される安全管理措置の負担の軽減だけでなく、情報が意図しない目的で利用されるリスクの軽減にもつながり、情報の提供者と利用者双方に有益なものとなっている。情報管理の基盤として、クラウドやコンテナの利用が進む中で、複数の機関が独自で管理している情報を組み合わせて解析を行う際の情報保護に関しても併せて検討が進められている。これらの技術は、本邦におけるバイオバンクや次世代医療基盤法の匿名加工医療情報作成事業者間の連携への応用も期待される。
情報の提供者が信頼して情報を提供できる環境を醸成するためには、漏洩や不正利用の抑止力となる規制の強化だけでなく、それらを事前に予防するための技術的措置も重要となる。ゲノム情報で検討されている研究利用における体制は、ゲノム情報だけでなく、他の医療情報においても応用可能な事例が多い。本邦の医療情報連携システムの整備に有益な示唆を与える先行事例として、本稿では単一機関でのデータのガバナンスに関して、Genomics Englandの事例を中心に紹介を行い、複数の機関によって管理される情報を組み合わせて利用する事例として、国家間でゲノム情報を利用することにより、ゲノム情報を用いた医療や医学の発展を目指す国際協力組織であるGA4GH(Global Alliance for Genomics and Health)によって開発されている研究体制の仕組みを紹介する。
医療情報連携の動向
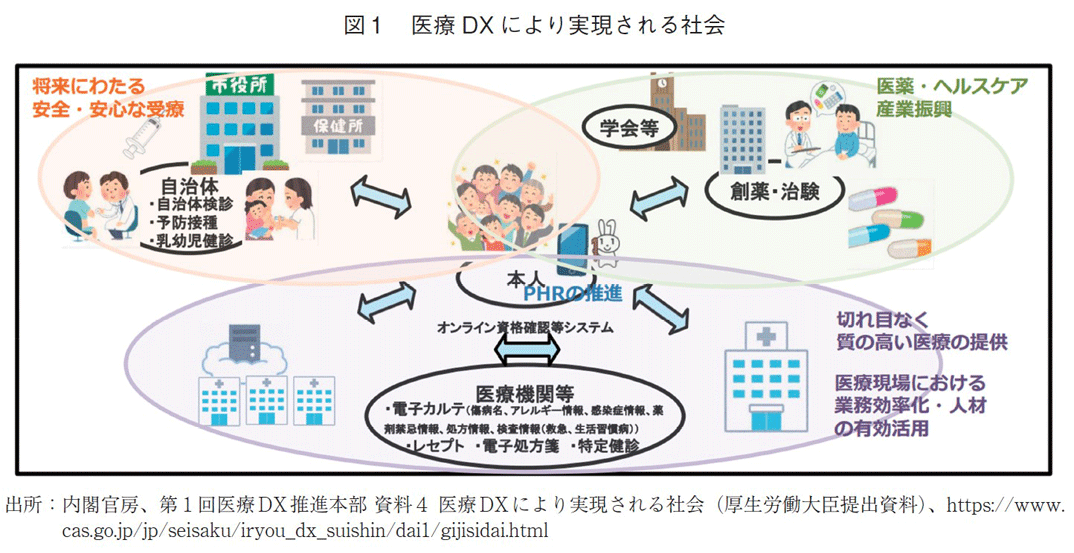
ゲノム情報に限らず、医療機関やバイオバンク等で管理される医療情報を他の医療機関や製薬企業と共有し、医療や創薬研究の効率化に繋げる動きは活発化してきており、日本製薬工業協会も本年4月に、一般の方に向けた医療情報の利用に関する啓発冊子を公開した4)。2022年10月に内閣に設置された医療DX推進本部においても、保健医療情報を共有することにより、創薬研究だけでなく、情報提供者本人に提供される医療の質向上を目指すことが、厚生労働省が提出した資料内で示されている5)(図1)。他国に目を向けても、新型コロナウイルスの感染拡大も要因となり、欧米を中心に、医療情報の共有が進められているが、2019年時点では医療機関が保有する情報の97%が利用できていないことが世界経済フォーラムから示されており6)、医療情報の利用による医療の質向上は、日本だけでなく世界中で注力されている課題となっている。医療情報の相互運用性の向上に関しては、主に情報の種別単位(FHIR-電子カルテ、DICOM-医用画像、IHE-システム連携など)で議論が進められてきた。これらの取り組みでは、主に自らの情報を提供した患者本人に対して効率的な医療を提供することを目的として、情報の構造定義や交換規約に関する整備が進んできたが、近年は研究開発などの二次利用への検討も始まっている。ゲノム情報の連携を促進しているGA4GHは、二次利用を進める過程で生じる問題を10個に分類しており、それぞれの課題解決に向けて活動を行っている7)。
医療情報の二次利用を阻む主な障壁
- 1.情報生成プロセスの統一性
- 2.相互運用性(データモデル・用語)
- 3.情報管理基盤
- 4.情報へのアクセス
- 5.情報の利用に関する同意取得
- 6.プライバシー・セキュリティの規制
- 7.情報提供者への透明性の確保
- 8.他国との情報共有(経験・信頼度)
- 9.情報共有・利用に対するインセンティブ
- 10.データの共有義務
本稿で紹介する個人情報保護のための技術的な措置は、主に「情報管理基盤」、「プライバシー・セキュリティの規制」の課題に対して有効な手段となる。情報管理に関する議論は、運用標準の統一とともに、医療情報の連携を阻む大きな障壁として存在する。英国では既に2013年から、医療機関が保有する情報を中央に設置したデータベースに集約を行い、医療情報の二次利用を行うCare.dataプロジェクトを行っていたが、個人情報保護の手続きが適切に実施されていないという理由で国民からの賛同が得られず、プロジェクトが廃止となった8)。近年では、当時と比較して医療情報の利用には一定の理解は進んでいるものの、昨年公表された米国医師会の調査においても、医療情報の二次利用に対する情報提供者の懸念は未だに大きいという結果が示されている9)。情報の利用を円滑に進めるためには、情報提供者の心理的な不安を軽減し、信頼を得るための仕組みの構築が必要であり、個人情報を技術的に保護する方法としてプライバシーテックが注目されている。
情報利用と保護の両立
はじめに、情報利用の体制整備から情報利用と個人情報保護との両立を目指している事例として英国のGenomics England(全ゲノムシーケンスを行う疾患コホート)における解析環境を紹介する。機微な情報を効率的に研究に利用していくためのフレームワークとして、Five Safes frameworkという考え方が存在する10)。英国の統計局が提案している考え方で、情報の利用時に考慮すべき要素を5つに分類している。英国の国民保健サービスであるNHS(National Health Service)を中心に健康保険分野の情報を利用した研究においても考慮されている。
Five Safes framework
- 1.Safe People(適切な利用手続き)
- 2.Safe Projects(適切な利用目的)
- 3.Safe Settings(不正利用の制限)
- 4.Safe Outputs(解析結果の管理)
- 5.Safe Data(情報の管理)
英国では、機微な情報を安全かつ確実な方法で研究利用するための環境をTRE(Trusted Research Environment)と呼び、機微な情報のプライバシーとセキュリティを保護しながら解析が実行できる環境の構築を目指している。TREではFive Safesのフレームワークを中心として個人情報保護の設計がなされており、NHSや保健省が関与するGenomics EnglandやOpenSAFELY(NHS患者記録の解析基盤)においても利用されている。
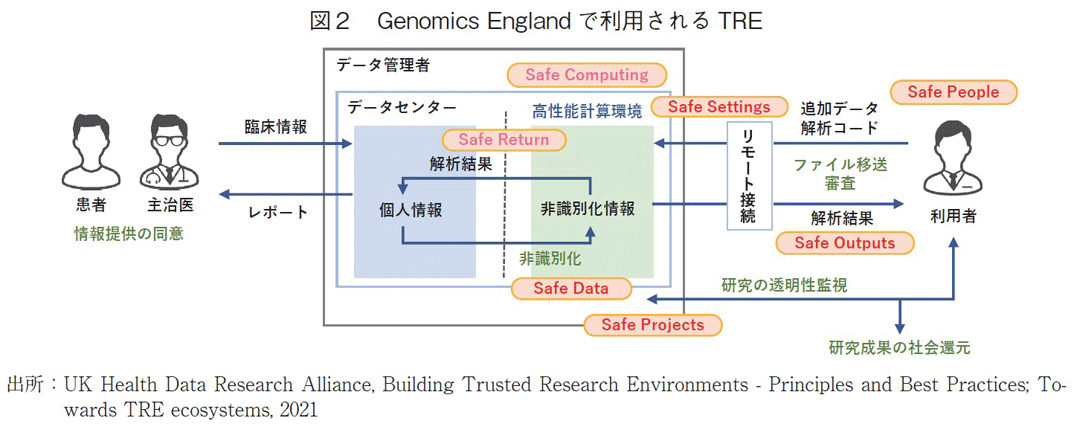
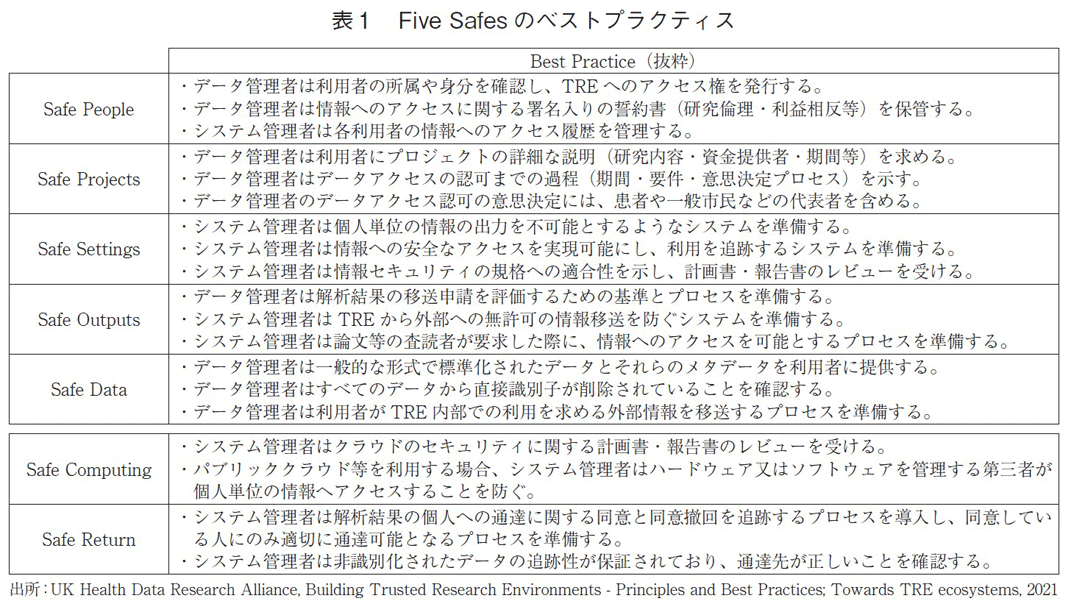
Genomics Englandでは、解析を実施する利用者にクラウド内に用意されたTREを提供しており、利用者は基本的にこの環境を利用して情報を扱うことになる11)。Genomics Englandでは、上記のFive Safesに加えて、2つの研究特異的な追加項目を含めて、7つの要素を意識した情報の管理がなされている。図2にTREの概要とFive Safesのフレームワークの適応が想定されるプロセスを示す。TREには、承認された利用者(Safe People)が、仮想デスクトップを介してのみアクセス可能となっており(Safe Settings)、利用者がアクセス可能な情報は非識別化された情報のみとなっている(Safe Data)。研究の内容は情報の管理者又は第三者によるレビューを受けることにより妥当性・透明性が確保される(Safe Projects)。TREから外部へは解析結果のみが持ち出し可能となっており、内容が確認されたのちに移送が可能となる(Safe Outputs)。Genomics Englandでは、他のプロジェクトと異なる特徴として、大規模情報を利用するためのパブリッククラウドの利用と、結果の一部をレポートとして主治医を介して情報提供者に提供しており、それぞれの過程においても個人情報の保護に関する考慮がなされている(Safe Computing、Safe Return)。英国の国立研究所の関連組織からFive Safesの各項目のベストプラクティスが公表されており、その中で重要と考えられる特徴を抜粋して表1に示す。
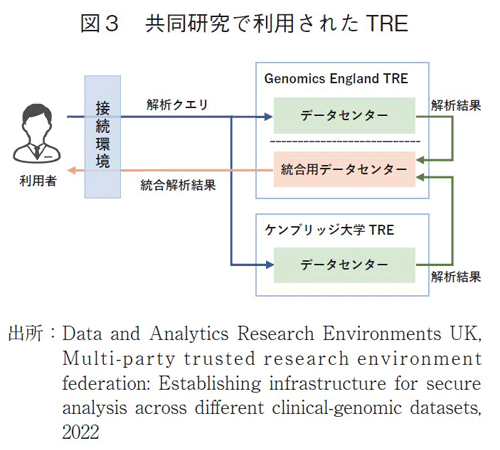
上記のようなTREを利用することは、単一の機関の中で解析を実施する際にはとても有効となるが、TRE外部への情報の持ち出しが厳格に制限されるため、解析結果の精度向上や検証を目的として、複数の機関で管理される情報を組み合わせた解析を実施することが困難となる。情報を医療機関の外部や、国外に持ち出すことが出来ない状況は、Genomics Englandに限らず、医療情報を解析する際には常に大きな制約となる。Genomics Englandでは、このような課題を解決するための実証研究として、ケンブリッジ大学の研究センターが保有する情報と組み合わせて解析を行うための体制を整備している12)。各機関から情報を持ち出さずに解析を行う方法は、Federated Approachと呼ばれており、解析クエリを各機関に共有し、各機関で実行した結果を集約することで統合した結果を得ることが可能となる(図3)。このプロジェクトでは時間的な制約により、統合用のデータセンターを、Genomics EnglandのTREに設置したが、本来は独立した環境に置くことが望ましいことが報告書の中で述べられている。以上のように、英国では、データの標準化が進むゲノムの分野を中心に、新たな情報利用の体制が採用され始めている。
情報利用の体制
Genomics Englandの事例において、クラウド環境やFederated Approachを用いた情報利用の体制を紹介したが、現時点で一般的に広く利用されている医療情報の共有方法は、ファイルサーバー又は電子記録媒体を介して情報自体を複製・配布する方法である。次世代医療基盤法においても、ガイドライン13)の中で「匿名加工医療情報取扱事業者に対する匿名加工医療情報の提供については、電気通信による方法や可搬記録媒体を用いる方法のほか、オンサイトで閲覧する方法も、想定される。」と記載があり、製薬企業が研究を行う際には、匿名加工医療情報は、製薬企業(匿名加工医療情報取扱事業者)が管理するストレージに保管されることが主な方法として想定されている。一方で、Genomics Englandの事例で紹介したように、クラウド技術やコンテナ型の仮想化技術の進展により、欧米を中心に情報利用の体制に変革がみられている。ゲノムや健康に関する情報の国際的な連携を促進するために設立された団体であるGA4GHが2021年に公表した論文では、3つの情報利用のアプローチが紹介されている14)(図4)。
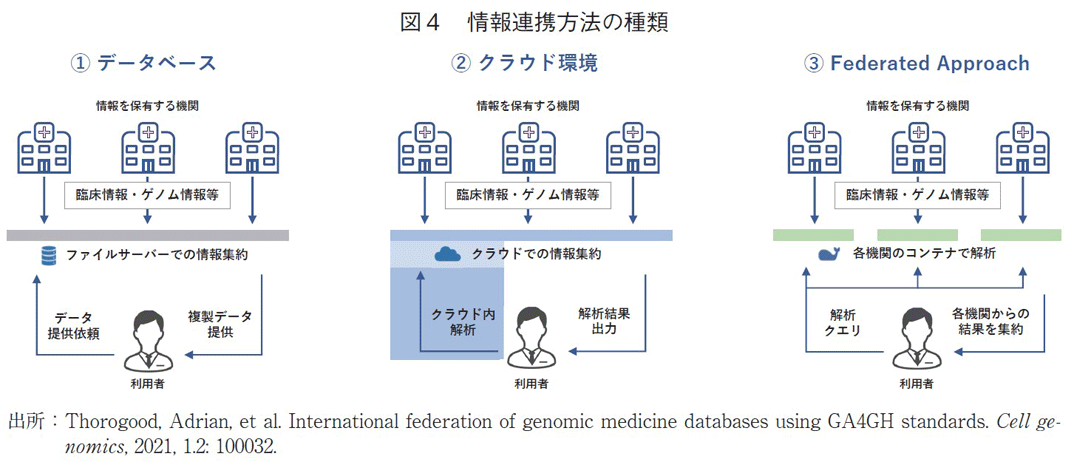
①に示す方法は、中央に設置したファイルサーバーにて、情報を一元化したのちに、集約した情報を利用者の基へ移送する方法である。上述の次世代医療基盤法においても想定されている方法で、認定医療情報等取扱受託事業者が情報の集約を担っている。②に示す方法は、利用者がクラウドに用意された解析環境を通じて、集約された情報にアクセスを行う方法である。解析前の医療情報を製薬企業が管理するストレージに保管しないという点で、上記の次世代医療基盤法ガイドライン中の「オンサイトで閲覧する方法」に類似する方法であり、機微な情報が利用者に渡ることがなく、より個人情報の保護に配慮した方法となる。③に示す方法は、解析前の医療情報を集約することなく、各機関の中で、利用者から提供された解析クエリを実行し、解析結果のみを集約する方法である。①と②に示す方法では、利用者が解析前の医療情報を閲覧可能な状況となってしまうため、さらに個人情報の保護を技術的に強化する方法として、近年注目され始めている。前述したGenomics EnglandのTREは②の方法で構築されており、ケンブリッジ大学との共同プロジェクトは③の方法に該当する。冒頭で紹介した、仮名加工医療情報を利用する際の懸念の多くは匿名加工医療情報を利用する際に採用していた①の方法に起因する懸念点が多く、②や③の方法では、情報の複製が不要となり、仮名加工医療情報の利用事業者が管理するストレージに情報が保存されることはなくなる。情報漏洩の観点だけでなく、情報の即時性や追跡性の観点においても①の方法と比較して優れている。
Genomics Englandの事例や、政策研ニュースNo.68で紹介したCOVID-19の事例からも、②のクラウド環境を利用した方法は欧米を中心に、既に様々な場面で利用が開始されており、本邦の東北メディカル・メガバンク機構においても遠隔地からの情報へのアクセスが可能となっている15)。③の分散型の情報利用の体制では、同時に全ての機関が管理する情報へアクセスすることが出来ないため、解析結果の精度の向上や、解析実施の体制整備など、現時点では解決すべき課題も多く存在しているが、Genomics Englandの事例のように実証研究が進められており、オランダを中心とするPersonal Health Trainの構想や16)、世界経済フォーラムから利用に向けた提言書が発出されており17)、研究途上であるものの実用化に向けて世界的に注目を集めている段階である。
ゲノム情報連携の動向
新たな情報利用の体制は、ゲノム研究の分野において現在最も実用化が進んでいる。ゲノム情報のデータベースは以前から情報の偏りが指摘されており、特に人種差に関しては、ゲノムワイド関連解析(GWAS:genome-wide association study)に利用されたゲノム情報は、ヨーロッパ系が78%以上を占め、アフリカ系は僅か2.4%程度であると推定されている18)。その結果、疾患の発症リスク等を予測するPRS(Polygenic Risk Score)のアフリカ系における予測精度が、ヨーロッパ系よりも約4.5倍低かったという報告もある19)。このような情報の不均衡は、新薬開発における効果の推定や、副作用リスクの推定にバイアスを生じさせる恐れがある。この問題を解消するためには、多様な民族から由来する情報を利用して解析を行う必要があるが、各国における民族の分布には当然偏りが存在しており、国際的な情報連携が必須となる。しかしながら、ゲノム情報の越境移転に関しては、厳しく制限している国も多く存在し、個人の情報を国外に出すことなく、精度の高い解析結果を得ることに対する需要は極めて高くなっている。ゲノム研究の分野では、前項で紹介した情報利用の体制を実践的に活用するためのプロジェクトも複数存在しており、それらの取り組みの一部を紹介する。
効率的な情報連携を目指すコンソーシアム
本稿では、情報の利用方法に焦点を当てているが、情報の利用を行う以前にも、情報提供者からの同意取得や、データ構造の標準化等の様々な解決すべき問題が存在する。これらの諸問題の解決に向けて活動を行っている組織は、OHDSI20)やCDISC21)、GO FAIR22)をはじめとして複数存在している。本稿では、その中でも本邦の研究機関も多数加盟しており、情報の利用方法の項でも紹介したように、ゲノム情報の連携において影響力の大きい取り組みを行っているGA4GHを紹介する。GA4GHは、前述のとおり、倫理的な配慮の下、国家間でゲノム情報を利用することにより、ゲノム情報を用いた医療や医学の発展を目指す国際協力組織である。56か国、680以上の組織が加盟しており、本邦においても、東北メディカル・メガバンク機構や、大規模なゲノム情報の統合利活用プラットフォーム(CANNDs)23)の構築を目指しているAMED(日本医療研究開発機構)等が加盟しており、本邦におけるゲノム情報の利用もGA4GHが提案するスキームを踏襲していくことが予想される。前述したGenomics Englandとケンブリッジ大学のプロジェクトにおいても、GA4GHによって開発された標準プロセスが採用されている。本邦でも、理化学研究所がGA4GHのプロジェクトとして、オーストラリアのQIMRベルクホーファー医学研究所とゲノム情報自体の授受を行うことなく、双方が保管するゲノム情報を利用してバリアントの評価を行ったことが発表された24)。これは、解析クエリを双方の組織で授受する方法によって実現されており、コンテナ型の仮想化技術を用いる方法(情報連携の③に示した方法)で実施された。
ゲノム情報の利用事例
本邦においても、AMEDや理化学研究所のような公的な組織が参画しているように、GA4GHで開発された技術や運用方針は各国のゲノムコンソーシアムで採用され始めている。本稿では、情報連携で先進的な取り組みを行っている事例を紹介する。
ELIXIR
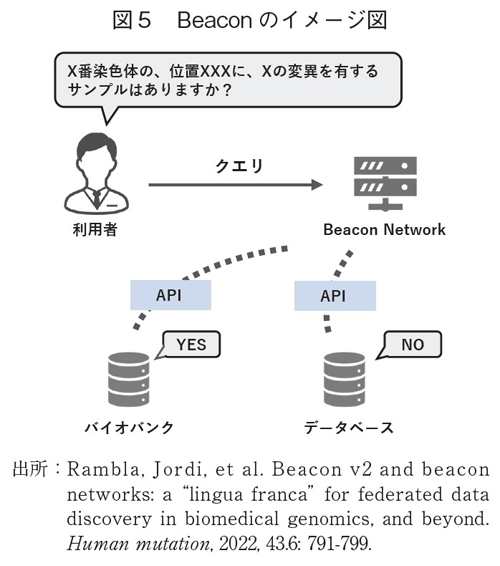
欧州において生体情報を横断的に利用可能とするための政府間組織であり、欧州の23か国の研究機関が参画している。ELIXIRでは、GA4GHと連携して参加機関が管理するゲノム情報を横断的に検索可能とするBeacon技術に関するプロジェクトが行われている。Beacon技術とは、各機関が管理するゲノム情報に含まれる特定の変異の有無に関する情報を提供するAPIであり、情報自体の共有を行うことなく、全参加機関の情報に対して検索が可能となるFederated Approachの一つである(図5)。このような横断検索を可能とする技術は、同一な構造の情報を複数の機関で分散して保持している際に有効となる。2021年時点では、42の機関がBeaconを利用しており、100万以上のサンプルから検索が可能となっている25)。現在でも、プロジェクトは進行中であり、ゲノム情報以外のフィルター条件の追加や、実際の情報へのアクセスを行う際の使用条件の確認等がBeaconを利用して可能となってきている。現在、Beacon技術は本邦のNBDC(National Bioscience Database Center)のデータベースにおいても採用されている。この技術は、ゲノムの変異だけでなく、疾患や使用薬剤にも応用可能であり、本邦におけるバイオバンクや次世代医療基盤法の匿名加工医療情報作成事業者の間の横断検索への応用が可能であると考えられる。
CanDIG
カナダにおいて医療情報を利用するためのプラットフォームであり、現時点でカナダにおいてゲノム情報を管理する5つのプロジェクトが参加している。カナダは複数の州からなる連邦国家であり、医療情報のプライバシー保護に関する規制は各州法に従う必要があるため、国内であっても個別情報の集約を行うことが困難である。この問題に対応するためにGA4GHで開発された分散型の情報利用の体制が採用されている。CanDIGでは、情報を保有する各参加機関が、自らが管理する情報の利用を完全に管理可能とするために、管理組織を設ける中央集権的なシステムの構築を避け、非中央集権型のモデルを採用した26)。独立した複数の事業者からなる本邦の匿名加工医療情報作成事業者においても、中央にプロジェクトを管理する組織を置くことが困難となる可能性があり、これらの取り組みは今後の運用の参考となり得る。このような非中央集権型の情報管理は、プラットフォーマーによる情報の独占などを避けるために近年注目されているが、管理組織を置かないことによって検討すべき事項も増加する。このような情報基盤を採用する際の留意点として、CanDIGでは以下の点が検討されている。
認証(AuthN:authentication)
認証とは、情報へのアクセスを行う個人が、本人であることを確認することを指す。中央集権型の方法をとる場合は、集約された情報を管理する機関において、一元的な管理がなされることが一般的であるが、非中央集権型の情報管理を行う場合は、情報アクセスの申請を受けた機関ごとに、情報利用を申請した個人の確認が必要となるため、本人確認の作業が、複数の機関で重複することとなる。そこで、GA4GHでは、GA4GH Passportという仕組みを導入し、OpenID Connect(承認サーバーの認証に基づいて、利用者の身元を確認する方法)により利用者のID情報を全ての参加機関で利用可能としており、CanDIGにおいてもこの認証方法が採用されている。
認可(AuthZ:authorization)
認可とは、認証された個人や組織が閲覧できる情報や、実行できる解析を決定することを指す。非中央集権型の情報管理を採用している場合は、情報へのアクセスに関する決定を外部の機関に委任することが難しい局面が多く、承認の最終決定は情報を管理する各機関が行うこととなる。CanDIG 内に保管されている情報の多くは、国家的なプロジェクトの一部であるため、各プロジェクトのデータアクセス委員会の決定とともに判断がなされている。
解析の流れ
非中央集権型の情報管理を採用している場合は、各機関において解析を実施する必要があるため、解析の実行方法の管理も必要となる。最も単純な方法は、利用者が全ての機関にクエリを個々に送付して結果を得る方法となるが、結果を集約する際に、それぞれの結果がどこの機関に由来する結果であるかが特定可能となり、情報漏洩に繋がるリスクが増加する。また、結果の統合を特定の機関が担うと、非中央集権型の情報管理を採用したメリットが低下してしまう。このような事項を踏まえ、CanDIGでは、これらの中間の考え方となるPeer-to-Peer型という方法がとられており、最初に解析を要求された機関が、他のすべての機関に解析の要求を行い、結果を統合することによって、解析の効率と個人情報保護の強化を行っている。
各機関での結果の共有
解析の流れの項目で触れたように、各機関内で解析を実行した場合、結果を統合する際に機関名と結果が紐づいてしまうと、情報漏洩に繋がるリスクが増加する。解析結果共有の際の技術的な対策として、差分プライバシーやマルチパーティー計算の利用が検討されている。CanDIGにおいては、現時点では参加機関が少なく、機関の間の信頼関係が強いためこれらの処理は採用していないが、今後プログラムが発展していくにつれて、導入の検討が必要となることが見込まれている。
情報へのアクセス管理
各機関に情報が分散していることにより、情報へのアクセス履歴も各機関において管理されることとなり、アクセス情報のサイロ化が生じることになる。アクセスの履歴は機密情報利用の追跡の観点と、情報の需要把握などにも利用可能な情報であり、参加する機関全体で履歴を管理できることが望ましいとされる。CanDIGでは、現時点では各機関で履歴を管理しており、機関の間で連携を行い全体の管理を行っているが、情報量や参加機関が増加するにつれてこのような管理手法の運用は不可能となるため、全体で情報へのアクセス履歴を管理できる方法への移行を検討している。
その他の取り組み
欧州やカナダだけではなく、その他の地域や疾患領域でGA4GHが開発した標準は利用され始めている。米国のNIH(国立衛生研究所)と英国のウェルカム・トラストはアフリカ科学アカデミーと共同で、アフリカ人集団に関する大規模なゲノム研究を行うH3Africaというコンソーシアムを設立し、情報の不均衡を解消する試みを行っている。その他にも、これまでに紹介した、欧州、カナダ、アフリカという大陸横断的な情報交換を目指すCINECAプロジェクトや希少疾患のゲノムプラットフォームであるMatchmakerでもGA4GHの標準が利用されている。
まとめ
本稿では、最も機微な個人情報として扱われているゲノム情報を中心に、情報連携の際に採用が検討される技術的措置を紹介した。今回紹介した方法は、個人情報を保護するために開発されているものであるため、情報の提供者を守ることが第一優先に考えられているが、情報を利用する製薬企業等の立場に立っても、個人情報の管理を容易にするものであり、製薬業界としても、医療情報の二次利用を行う際に導入を促進していく必要があると考える。特に、次世代医療基盤法の改正に伴って認定利用事業者に求められる、より強固な情報管理への対応として、Five Safes frameworkとクラウド環境を組み合わせたGenomics Englandの構造は参考となる点が多いだろう。バイオバンクでは、東北メディカル・メガバンク機構では、既に遠隔地からの情報へのアクセス、スーパーコンピュータの利用が可能となっており、他のバイオバンクにおいても解析環境の整備は進んでいくことが見込まれる。さらに、機微な情報を機関の外部に提供できないことにより、情報の統合が困難になっている状況は、本邦におけるバイオバンクや次世代医療基盤法の匿名加工医療情報作成事業者においても、本稿で示した事例と類似した事象が発生しており、利用者側からはどの事業者が求める情報を保持しているのかが認識しづらい現状がある。バイオバンクに関しては、AMEDが国内の大規模ゲノム情報を検索可能とする計画において、クラウドを利用した解析環境を構築することが公表されているが27)、統合検索環境で各バイオバンクが保有している情報に関して、どこまで詳細な情報を利用した分析が可能となるかに応じて、情報アクセスの構造を検討する必要がある。次世代医療基盤法に関しても、将来的に仮名加工医療情報作成事業者が増加していくと、仮名加工医療情報作成事業者の間での情報連携が求められることが想定される。機微な情報を扱うために丁寧に検討が行われているゲノム分野での情報連携の構造は、本邦の医療情報の活用においても応用可能な視点が多く存在しており、法的措置の強化だけではなく、新たな技術を活用した情報保護の強化と情報活用の推進に関する議論も進んでいくことが期待される。
-
1)
-
2)
-
3)
-
4)
-
5)
-
6)
-
7)Rehm, Heidi L., et al. GA4GH: International policies and standards for data sharing across genomic research andhealthcare. Cell genomics, 2021, 1.2.
-
8)Limb, Matthew. Controversial database of medical records is scrapped over security concerns. BMJ: British MedicalJournal, 2016, 354.
-
9)
-
10)Ritchie, Felix. Secure access to confidential microdata: four years of the Virtual Microdata Laboratory. Economic &Labour Market Review, 2008, 2: 29-34.
-
11)UK Health Data Research Alliance, Building Trusted Research Environments - Principles and Best Practices;Towards TRE ecosystems, 2021
-
12)Data and Analytics Research Environments UK, Multi-party trusted research environment federation: Establishinginfrastructure for secure analysis across different clinical-genomic datasets, 2022
-
13)内閣府、医療分野の研究開発に資するための匿名加工医療情報に関する法律についてのガイドライン、2023
-
14)Thorogood, Adrian, et al. International federation of genomic medicine databases using GA4GH standards. Cellgenomics, 2021, 1.2: 100032.
-
15)
-
16)Beyan, Oya, et al. Distributed analytics on sensitive medical data: the personal health train. Data Intelligence, 2020,2.1-2: 96-107.
-
17)The World Economic Forum, Federated Data Systems: Balancing Innovation and Trust in the Use of Sensitive Data,2019
-
18)Atutornu, Jerome, et al. Towards equitable and trustworthy genomics research. EBioMedicine, 2022, 76: 103879.
-
19)Martin, Alicia R., et al. Clinical use of current polygenic risk scores may exacerbate health disparities. Naturegenetics, 2019, 51.4: 584-591.
-
20)
-
21)
-
22)
-
23)
-
24)Casaletto, James, et al. Federated analysis of BRCA1 and BRCA2 variation in a Japanese cohort. Cell genomics, 2022,2.3: 100109.
-
25)Harrow, Jennifer, et al. ELIXIR-EXCELERATE: establishing Europe’s data infrastructure for the life science researchof the future. The EMBO Journal, 2021, 40.6: e107409.
-
26)Dursi, L. Jonathan, et al. CanDIG: Federated network across Canada for multi-omic and health data discovery andanalysis. Cell Genomics, 2021, 1.2: 100033.
-
27)

