Points of View COVID-19の事例から見る医療記録統合の動向
医薬産業政策研究所 主任研究員 岡田法大
近年、実臨床の中で取得される医療記録等の情報を研究開発や疫学調査へ利用するための検討が医薬品業界においても活発に行われている。これらの情報を利用する際には、推定精度を向上させるための症例数の確保や、結果の外的妥当性を向上させる目的で、単一の医療機関で得られる情報のみを用いて解析を行うのではなく、複数の医療機関で収集された情報を統合して解析を行うことが望ましい場面が多い。一方で、統合した解析を行うために、患者の情報を医療機関の外部に提供することは、本邦における個人情報保護法や米国におけるHIPAA(Health Insurance Portabilityand Accountability Act)等の各国の法律で規制されており、医療機関を横断して患者単位の医療情報を統合することは容易ではない。この問題に対して、本邦においても次世代医療基盤法が施行され、要配慮個人情報の認定事業者への提供がオプトアウトにより可能となったように、医療情報の利用促進を目指した施策が講じられているものの、本人通知の際の医療機関の負担や認定事業者間の連携等、依然として多くの障壁が残されていることは、昨年末から開催されている次世代医療基盤法検討のワーキンググループ内での議論1)でも窺うことができる。解決に向けては、法整備に関しての議論が注目を集めることが多いが、昨今では、医療記録を研究利用することにより得られる利益を保ちつつ、個人情報漏洩によるリスクを軽減するために、技術的な側面からも様々な情報統合のアプローチが開発され、実用化に向けた検討がなされている。本稿では、多様なアプローチが実践された新型コロナウイルス感染症(COVID-19)における、電子カルテを中心とした医療記録(EHR:Electronic Health Record)の統合を事例として、医療情報の統合の在り方に関して考察したい。
医療情報の統合方法
医療機関が保有する情報を統合したデータベースを研究へと利用する検討は、各国の政府が主導するものや、国境を越えた統合を目指すものなど、多様な疾患を対象に世界的に行われている。情報の統合は、主に以下の3種類の方法を用いて実現されている。(図1)
-
①個別症例情報の集約
中央の統合環境から各医療機関が保有する患者単位の情報にアクセスが可能な状態とする、又は複製した患者単位の情報を中央のストレージに集約した後に、統合された環境において解析を実施する方法。
-
②医療機関で要約された情報の集約
各医療機関において、規制上外部提供可能となる粒度まで個人情報を要約し、要約された情報のみを中央のストレージに集約した後に、統合された環境において解析を実施する方法。
-
③医療機関で実行された解析パラメータの共有
予測・分類モデルを構築する際に利用される方法で、各医療機関において、保有する情報を用いてモデルのパラメータの更新を行い、パラメータを中央の統合環境に集約し、集約したパラメータから得られた情報を再度、各医療機関に戻してパラメータ更新を繰り返し行うことにより、すべての医療機関のデータを用いて調整した共有のモデルを構築する方法。Federated learningやSplit learningと呼ばれるフレームワークが考案されている。
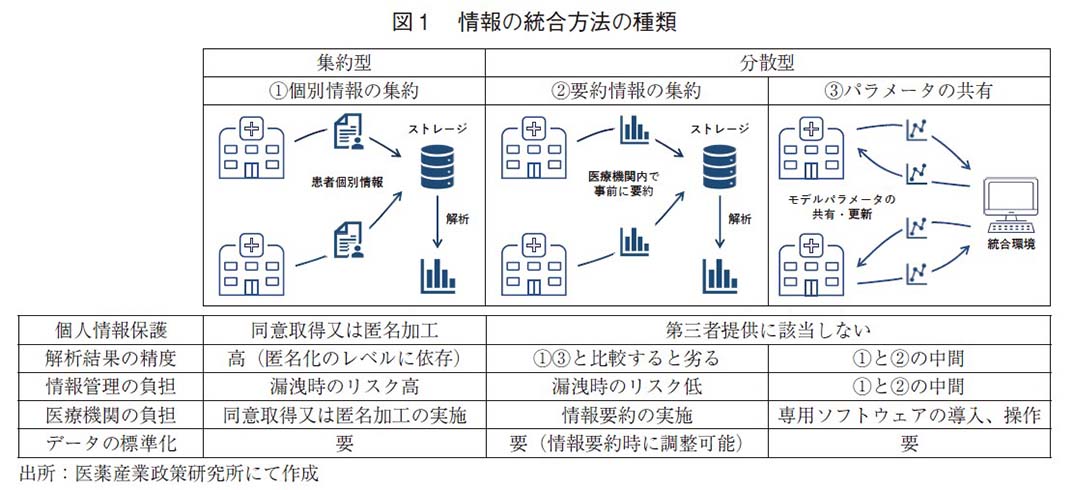
一般的に用いられる統計処理は、基本的に全ての情報が一箇所のストレージに格納されていることを想定しているため、最も解析に適した情報の統合方法は、①の個別症例の情報を一箇所に集約する方法である。この方法では、多くの場合が個人情報の第三者提供に該当するため、情報提供者からの同意取得、又は提供する情報の匿名加工が求められる。解析の観点のみを考慮すると、全ての患者から同意を取得することが望ましいが、次世代医療基盤法で求められているオプトアウトに関する通知に対しても、要件緩和の要求が出ている現状2)から、同意の取得は医療機関において大きな負担となる。加えて、同意の有無によって生じる患者選択のバイアスが解析に与える影響に関しても検討を行う必要が生じる。情報管理の観点では、第三者として情報を受領する立場側の視点でも、個人情報又は匿名加工情報を直接扱う必要が生じるため、情報の漏洩が生じた際のリスクは大きくなり、情報管理のコストは増大する。また、匿名加工情報には再識別リスクへの対応や匿名化による情報の損失等の問題も存在する。このように、個別症例の情報を集約する①の方法は、複数の懸念点が存在するものの、構造上は最も単純であり、解析上で有利な点が多いため、現在でも最も多く利用されているデータの統合方法の一つである。
個別症例の情報の集約と共に多く利用されている方法として、②の医療機関内で情報の要約を行い、要約の結果を中央のストレージに集約する方法がある。最も単純な例として、厚生労働省が実施する季節性インフルエンザの流行状況の定点調査がある。定点調査では、医療機関単位で性別、年齢階層別に集計された患者数を保健所に提出し、各医療機関の集計結果を基に都道府県単位で感染者数が再集計される。このように、必要とする情報が患者数等の集計値であれば、医療機関における解析の負担は増すものの、個別症例の情報を統合する必要はなく、情報の管理も比較的容易となる。
最後に、③の解析パラメータの共有を繰り返すことで、個別症例の情報の共有を行わずに予測・分類モデルを作成する方法がある。この方法は、2016年にGoogle社から発表されたFederated learningと呼ばれるフレームワークを皮切りに、実用化に向けた研究が多く行われている3)。②の要約情報を集約する方法の派生であり、同様に、患者単位の情報を第三者に提供する必要がない。単一の医療機関でモデル構築を行う場合と比較して、外的妥当性が高いモデルを構築することが可能となる。
COVID-19での情報統合方法の事例
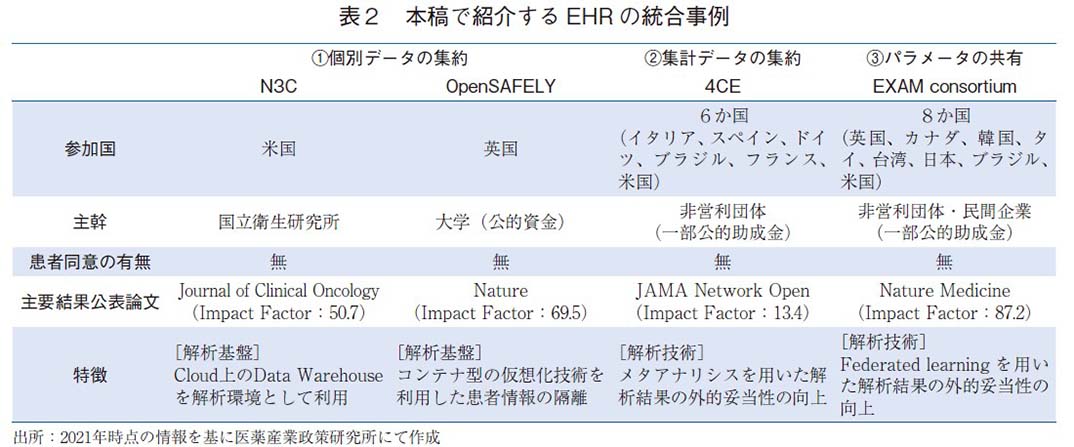
本稿では、最新の医療情報の統合方法の潮流を理解するために、全世界で多様なデータベースが作成されているCOVID-19におけるEHRの統合方法を基に、統合方法の特徴と個人情報保護への対応方法を整理する。①の個別症例の情報を集約する方法の事例として、米国と英国(イングランド)の政府機関が援助して作成されたデータベース、②の要約した情報を集約する方法の事例として、COVID-19の国際的なEHRデータとしては最大規模のコンソーシアム、③の解析パラメータを共有する方法の事例として、ハーバード大学医学部の医療機関とNVIDIA社が行った研究をそれぞれ紹介する。
①-1 National COVID Cohort Collaborative(N3C)4)
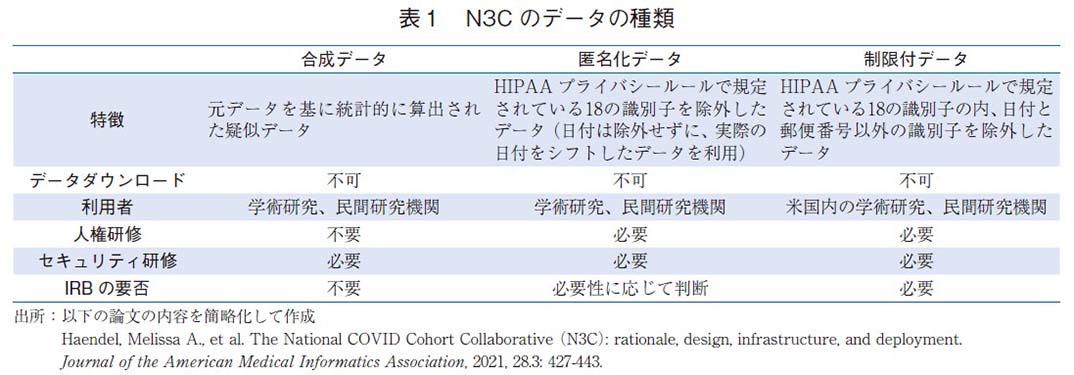
米国の国立衛生研究所の研究機関の一つであるNCATS5)が主導するプロジェクトで、COVID-19患者のEHRの統合データベースを研究目的で構築している。データベースには米国の50以上の医療機関から収集された640万人以上のCOVID-19患者の情報が含まれている(2021年11月時点6))。データの集約に際しては、特例措置として患者からの同意取得が免除されており、HIPAAに基づいて個人識別に繋がる情報を削除し、匿名化を行うことにより研究利用が可能となっている7)。HIPAAで規定されている削除すべき18の識別子(名前、住所、日付、電話番号、FAX番号、メールアドレス、社会保障番号、カルテ番号、保険番号、口座番号、各種免許書番号、車両番号、デバイス識別番号、URL、IPアドレス、生体認証識別子、本人写真、その他の固有の識別番号)の内、N3Cではパンデミックの流行追跡のために、住所情報(郵便番号)と日付の情報を有しているため、解析目的に応じて提供されるデータセットのセキュリティレベルを分類し、レベルに応じてIRBでの承認や国外の機関からのアクセス制限等の追加の対策がとられている(表1)。情報保護の観点では、技術的な対応もとられており、データへのアクセスは米国政府の要件定義を満たしたGov-Cloud(Amazon Web Services)上に用意された解析プラットフォーム(Palantir Foundry)内に制限されており、情報へのアクセスや結果の出力の履歴が全て中央で管理される。解析環境にはPythonとRの他にApache SparkやBIツールも用意されている。
①-2 OpenSAFELY8)
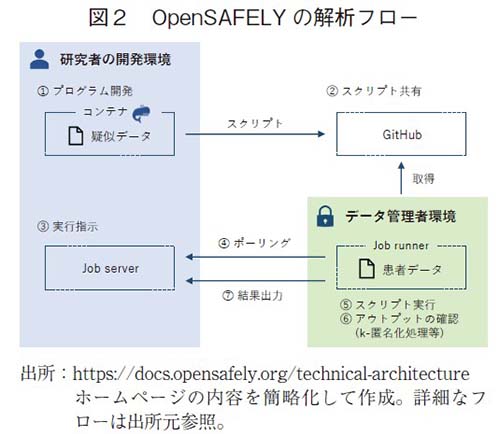
イングランドの国民保険サービス(NHS England)の支援を受け、オックスフォード大学が主導して開発したEHRの解析プラットフォームで、COVID-19の流行を機に構築された。電子カルテベンダーから患者の記録が提供されており、COVID-19患者以外の情報も含めて英国の約5800万人の患者記録が含まれている9)(2021年10月時点10))。英国では患者情報を用いたCOVID-19研究に関する特例の通知11)が出されており、データの集約に際して、患者から研究利用に関する同意は取得しておらず、収集された情報はGDPR(General Data Protection Regulation)とDataProtection Actに基づいて保護され、ハッシュ関数を用いて仮名化される。情報保護に関する技術的な側面では、データを中央で一元管理することにより、情報へのアクセスや結果の出力の履歴が全て管理される点はN3Cと同様だが、更なる特徴として、データアクセスの承認を得た研究者も、実際のデータにアクセスすることが不可能な構造をとっている。これは、コンテナ技術やコード管理システム等を用いることによって実現されており、研究者が一度もデータを閲覧することなく、出力を得ることが可能となる(図2)。ソースコードもGitHub上で公開されており、解析内容の透明性確保の観点も含めて個人情報保護を意識した先進的な設計がなされている。
② Consortium for Clinical Characterization of COVID-19 by EHR(4CE)12)
EHRをはじめとする医療データの共有、統合、標準化を目指す組織が中心となった国際的なコンソーシアムであり、6か国の315の医療機関で構成されている。データベースには約8万人の患者記録が含まれている(2021年10月時点13))。参加している医療機関が組織内で解析を実行し、その集計結果を中央のストレージで管理する仕組みとなっており、組織の外部に提供されるデータに個人情報は含まれないため、患者から研究利用に関する同意は取得していない。参加している医療機関は、医療データの標準化プラットフォームを導入しており、同様のデータ構造で情報を管理しているため、各医療機関において、中央で作成された共通の解析用スクリプトを実行するだけで解析が完了する仕組みとなっている。
③ EXAM(EMR CXR AI Model)consortium14)
ボストンを拠点とする病院ネットワークであるMass General BrighamとNVIDIA社を中心とするFederated learningを研究する国際的なコンソーシアムであり、8か国の20の医療機関からなる。2021年9月に結果が公表された研究には約1万6千人の患者記録が利用されている15)。②の方法と同様に、組織の外部に提供されるデータに個人情報は含まれないため、患者から研究利用に関する同意は取得していない。個別症例に関する情報の授受を行わない②と③の統合方法は、情報の越境移転を伴う複数の国で構成されるプロジェクトで採用される事例が多い。
本稿で紹介するEHRの統合事例について、表2に概要を記す。
得られる成果から見るデータベースの特徴
情報をどのように統合するかは、情報の利用目的を明確にした後に検討されることが望ましい。しかしながら、医療記録の集積には数年から数十年の単位を要するため、長期的な視野を持ち、同一のデータベースを幅広い研究に転用可能とするために、情報の統合方法の議論が利用目的の決定に先行して行われる場合が多い。本稿では、立ち返って利用目的に応じた適切なデータの統合方法について考察したい。
EHRを用いた解析の妥当性は、アウトカムの定義、未測定の交絡因子、欠測のメカニズム等の観点で多様な議論があるが、本稿では個別症例の情報を集約した状態においては、求める解析が実施可能となるという仮定で考察を進める。今回事例として挙げたEHRから得られる情報を利用して実施される解析は、大別すると以下の3種類に分類することができる。
- A.要約統計量の算出
- B.分類・予測モデルの構築
- C.治療・薬剤間比較
A. 要約統計量の算出
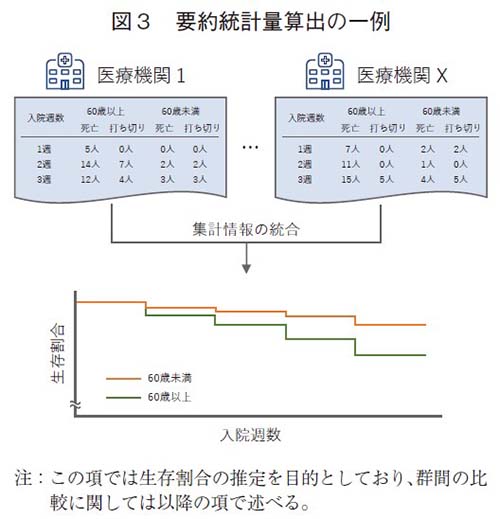
COVID-19の事例では、PCR検査の陽性者数や死者数、対象患者の年齢や性別等の背景情報の集計が該当する。このような解析では、要約された情報を集約した場合でも、個別症例の情報を集約した際とほぼ同じ精度で結果を得ることが可能となる。例えば、入院後の転帰情報に関して、Kaplan-Meier曲線を作成し、生存時間の推定を行いたい場合も、集計データを加工することにより可能であり、個別症例の情報を集約することは必須ではない(図3)。
個人情報保護の観点では、患者から同意を取得した場合を除き、匿名化された情報を統合する際には、情報の再識別リスクを軽減するために特異な記述の削除(k-匿名化等)の加工が施されることが一般的である。この処理が必要な場面では、個別症例の情報を集約する際でも同様に実施されるため、得られる結果に与える影響の違いは無い。一方で、医療機関で集計を行った結果を組織の外部へ提供することにより、個人単位での変数間の関連を削除することが可能となるため、複数の情報を組み合わせることによる個人の再識別のリスクを減少することは可能となる。このように、各医療機関での集計時の負担が必要となることを除けば、個別症例の情報を集約する方法に対する優位性は大きく、要約統計量の算出を行う際は、個人情報を集約しない構造が望まれる。しかしながら、医薬品産業においては、要約統計のみで解決する課題はほとんど存在しないため、以降の項目も併せて検討が必要となる。
B. 分類・予測モデルの構築
医療記録を用いた解析において最も需要が大きい解析の一つとなるのが分類・予測モデルの構築である。医薬品産業においても、薬剤の効果の予測は然ることながら、有害事象のリスク評価時の交絡調整やバイオマーカーの探索等で広く用いられている。解析の精度の観点から見ると、個別の症例の情報を集約することが最も望ましいが、個人情報保護に考慮しつつ、個別症例の情報を集約した際の精度に近づける解析手法も多く研究が行われている。本項では、COVID-19の重症化・死亡のリスクを予測するモデルの構築に焦点を当て、各データ統合方法でのアプローチを紹介する。
①の個別症例の情報を集約する方法では、情報が集約された環境において、研究者がモデルを構築することで目的が達成される。米国のN3Cと英国のOpenSAFELYでは、それぞれ前述したように、指定された環境での解析や、実際のデータが閲覧できない中での解析等の制約が存在しているが、解析環境が綿密に設計されていれば、目的達成への大きな障害とはならない。N3Cのデータを用いた研究では、COVID-19の重症度を患者背景(年齢、性別、合併症等)や臨床検査値の値を用いて重症化等を予測するモデルが構築されている16)。計算を中央で一括して実行できるため、探索的に多様なモデルを適用し、最適なモデル選択に関する検討も実施されている。N3Cの解析環境には分散処理のフレームワークも用意されており、データを一箇所に保持していることの利点を活かした研究が進められている。OpenSAFELYでも同様に、患者背景や併存疾患から死亡リスクを予測するモデルの構築がされている17)。
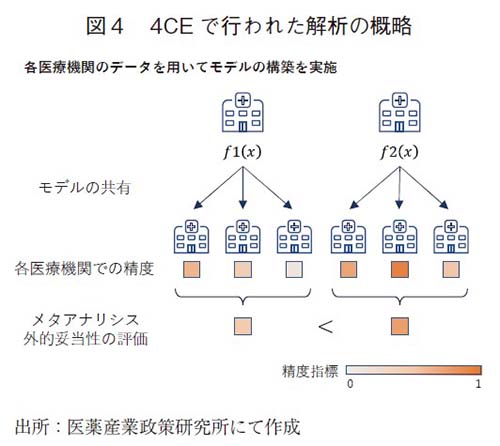
一方で、②の要約された情報のみを集約する方法でモデルを構築する場合、モデルパラメータの調整に他の医療機関の情報を利用することが難しい。4CEでは、COVID-19の死亡リスクを患者背景や臨床検査値の値を用いて予測するモデルが構築されているが、そこでは、各医療機関が有する情報を用いて、それぞれの医療機関でモデルの構築を独自に行い、構築されたモデルを別の医療機関のデータに適用するアプローチがとられた18)。作成されたモデルを他の医療機関で検証し、メタアナリシスの手法を用いて結果を統合することにより、国単位や医療機関単位でモデルの特徴を整理しながら外的妥当性を評価する試みを行っている(図4)。このようなアプローチが実施できている背景には、4CEの母体がEHRデータの標準化を目指すコンソーシアムであり、予め共通のデータ形式(CDM:Common Data Model)を用いてデータが管理されていたことの貢献が大きい。本手法でも、アルブミンの低値やリンパ球数の低値などがCOVID-19の予後に影響することが検出された。
一方で、②の要約された情報を集約する方法で紹介したメタアナリシスでは、複数の医療機関の情報を用いてモデルのパラメータの更新を行うことが出来ないため、モデルの精度を最大限に向上するという側面では限界があり、その欠点を補うために研究されているのが、③の解析パラメータの共有を繰り返す方法である。EXAM consortiumの研究では背景情報、臨床検査値に加えて胸部X線画像から予後(必要酸素投与量)の予測を行っており、この方法で構築されたモデルは、各医療機関が保持する単独の情報を用いて構築したモデルと比較して、全ての医療機関において優れた精度のモデルとなったことが報告されている15)。解析パラメータを複数回共有することは、要約された情報を一回のみ共有する②の方法と比較すると、パラメータ更新のための通信を傍受されていた場合の個人情報の識別リスクが上昇する。そのため、EXAM consortiumでは、差分プライバシー3)を用いた識別リスクの軽減についての研究も行っており、個人情報保護の観点でより安全に、求める成果を得るための試行がなされている。
C.治療・薬剤間比較
通常、医薬品の有効性や安全性を検証する際には、他の治療との比較を行う臨床試験が実施される。これは、無作為化と盲検化により、比較する群間で介入以外の因子の影響を排除するためであるが、データベースを利用した治療の比較においては、これらの処置を施すことが不可能であるため、比較可能性の担保、主に交絡の調整が最も重要な論点となる。現時点で、個別症例の情報を集約せずに、治療間の比較が行われた事例が少ないため、本項ではCOVID-19での事例ではなく、将来的に個別症例の情報を集約することなく研究が可能となるかを考える。想定される状況としては、比較したい双方の群の情報が同一のデータベース内に存在する場合と、単群で実施した臨床研究のコントロール群としてデータベースを用いる場合(外部対照群としての利用)が挙げられる。交絡因子の調整に最も広く用いられている傾向スコアによる調整を想定すると、同一のデータベース内に比較を行う両群のデータが存在する場合は、分類・予測モデルの構築の項で紹介した方法で、ロジスティック回帰等を用いることで傾向スコアの算出が理論上は可能となる。医療機関横断的にマッチングを行う際には、傾向スコア自体を第三者に共有する必要があり、個人情報の再識別リスクに細心の注意を払うのであれば、傾向スコアの算出パラメータを知る第三者への共有の回避や、逆確率による重みづけ等での調整を検討することが望まれる。一方で、外部対照群として用いる場合には、各医療機関が保有する情報が臨床研究で得られた情報の対照となるため、比較を行う治療の群が、医療機関側と臨床研究側のデータで完全に分離されることとなる。このような状況では、各医療機関内でパラメータの更新を行い、傾向スコアの推定精度を向上させることは、現行の技術では困難となることが想像される。
展望
解析における利便性の観点では、個別症例の情報を一元管理することが望まれるが、個人情報保護の観点とそれに対応する技術の発展により、世界的に情報の統合は個別症例の情報を秘匿して解析を行う方向に進んでいることが窺える。本稿で紹介した米国のN3Cにおいても、個別症例の情報を一元管理しない方法を選択することも当初検討していたが、プロジェクトが複雑になることを懸念して、現時点では断念したことが論文に記述されている19)。個別症例の情報が必要となる場合においても、クラウド環境においてデータ自体と解析における処理を制御する設計を構築することで情報漏洩のリスクを軽減することが可能となった。情報反映の即時性の観点でもデータを一元管理することは有効であり、本邦において実施されている匿名加工情報の移送や、オンサイトセンターでの解析等も順次、クラウド等の仮想化サーバー上での解析に転換していくことが望まれる。また、今回紹介した全ての事例は、情報の統合方法に関わらず、CDMを用いてデータの形式が類似していたことにより実現されており、本邦においても情報の標準化には引き続き注力していく必要がある。データベースから得られる結果の精度と個人情報保護の強度はトレードオフの関係にあり、個別症例の情報を収集せずに実行できる解析の幅が広がりつつある現在、解析の目的や求める精度に応じて最適なデータの統合方法を決定することはより重要になってくるだろう。
-
1)
-
2)
-
3)医薬産業政策研究所、「プライバシー保護技術に関する動向と医療ヘルスケアデータの利活用における示唆」、政策研ニュースNo.61(2020年11月)
-
4)
-
5)
-
6)Pfaff, Emily R., et al. Synergies between centralized and federated approaches to data quality: a report from the national COVID cohort collaborative. Journal of the American Medical Informatics Association, 2022, 29.4: 609-618.
-
7)NIH のIRB から、Federal Policy for the Protection of Human Subjects (‘Common Rule’)に準拠した同意の免除を受けた。
-
8)
-
9)実際には2社の電子カルテプロバイダーの情報を分散型の統合方法で扱っているが、医療機関からの視点で述べている本稿では、個別症例のデータを統合したデータベースとして取り扱う。
-
10)Walker, Alex J., et al. Clinical coding of long COVID in English primary care: a federated analysis of 58 million patient records in situ using OpenSAFELY. British Journal of General Practice, 2021, 71.712: e806-e814.
-
11)
-
12)
-
13)Weber, Griffin M., et al. International changes in COVID-19 clinical trajectories across 315 hospitals and 6 countries: retrospective cohort study. Journal of medical Internet research, 2021, 23.10: e31400.
-
14)
-
15)Dayan, Ittai, et al. Federated learning for predicting clinical outcomes in patients with COVID-19. Nature medicine, 2021, 27.10: 1735-1743.
-
16)Bennett, Tellen D., et al. Clinical characterization and prediction of clinical severity of SARS-CoV-2 infection among US adults using data from the US National COVID Cohort Collaborative. JAMA network open, 2021, 4.7: e2116901.
-
17)Williamson, Elizabeth J., et al. Factors associated with COVID-19-related death using OpenSAFELY. Nature, 2020, 584.7821: 430-436.
-
18)Weber, Griffin M., et al. International comparisons of laboratory values from the 4CE collaborative to predict COVID-19 mortality. NPJ digital medicine, 2022, 5.1: 1-8.
-
19)Haendel, Melissa A., et al. The National COVID Cohort Collaborative(N3C): rationale, design, infrastructure, and deployment. Journal of the American Medical Informatics Association, 2021, 28.3: 427-443.

